2.2. Técnicas de secuenciación
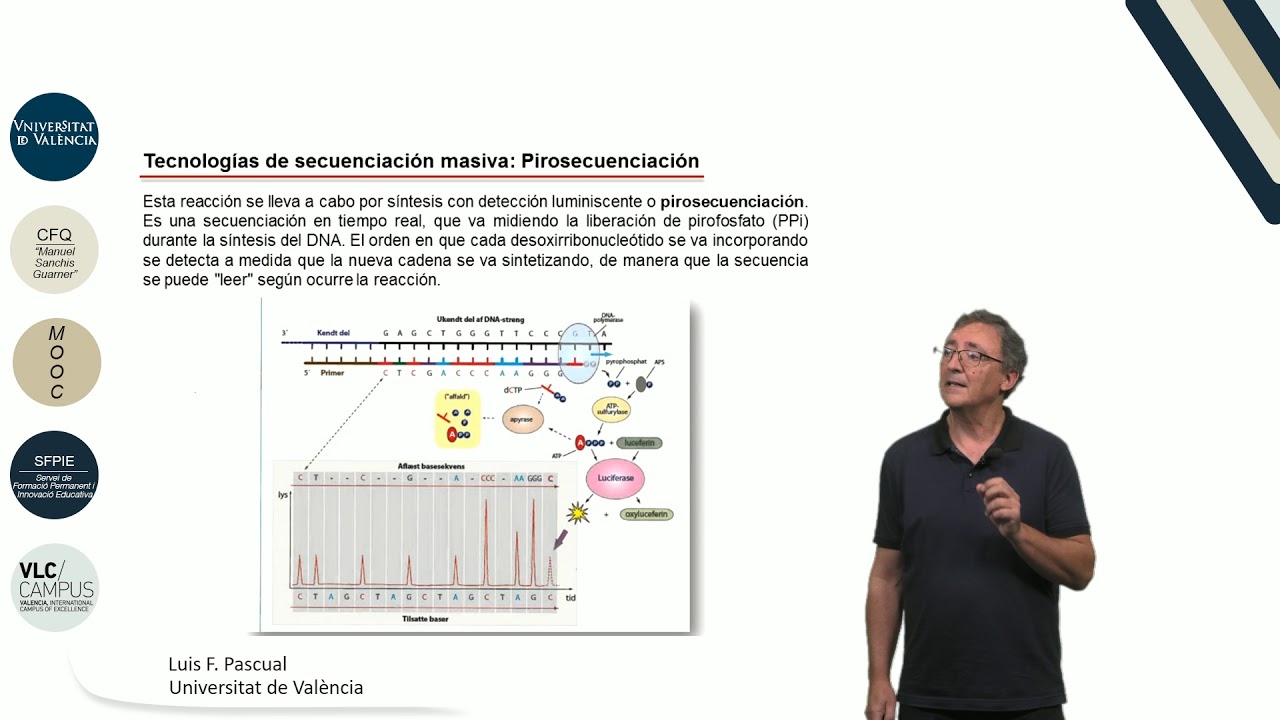
[Música] hola soy luis pascual y voy a hablaros de técnicas de secuenciación la secuenciación es el proceso mediante el cual se determina el orden preciso de las bases nitrogenadas de un fragmento de una cadena de adn aunque en el año 65 ya se consiguió la primera secuencia de un gen no fue hasta la instrucción de las tecnologías del dna recombinante que se posibilitó abordar el reto y desarrollar métodos que fuesen realmente efectivos para la secuenciación de genes de hecho fue el equipo dirigido por frank sánchez y el que desarrolló el método conocido como secuenciación mediante vídeo shocks y nucleótidos o método de sánchez que vino a convertirse durante muchos años en el procedimiento estándar para secuenciar cualquier fragmento purificado de adn este método se basa en la síntesis enzimática de la cadena complementaria aquella que se quiere secuenciar los elementos de la reacción enzimática de polimerización son el dna molde que va a ser que haya sido clonado en un vector apropiado un oligonucleótidos que se empareja con este y actúa de cebador los cuatro nucleótidos trifosfato uno de los cuales están marcados con un flúor o cromo por radio activamente y un adn a polimerasa este cóctel se introduce en cuatro reacciones en paralelo en cada una de las cuales se prepara o se añade un di de shocks y núcleo kilo diferente el directorio nucleótido de adenina de timina citosina o de guanina como aparece en la imagen en cada una de estas reacciones lo que ocurre es que la secuencia molde que la tenemos en la imagen de la izquierda abajo la secuencia molde es leída y es polimerizado por el encima y va introduciendo el nucleótido hasta el momento en el que introducen el vídeos oxido correspondiente a ese tubo de reacción en este momento la polimerización se bloquea y se genera un fragmento de un tamaño determinado que viene condicionado por la introducción de este núcleo en el caso del que aparece la imagen sabemos que es un nucleótido de adenina el resultado de la reacción es por tanto una serie de fragmentos de distintos tamaños todos acaban todos acabados en una incorporación de un vídeo socks ido de adivina que luego al ser separados en un gel de electroforesis dan toda una serie de fragmentos que al ser leídos pueden dar pie a la reconstrucción de la secuencia de la secuencia molde que queríamos que queríamos conocer el método este de secuenciación de as ángel fue mejorado con el tiempo utilizando por una parte nuevas polimerasa las conocidas como sequen asas combinando la reacción de secuenciación completa y áreas asimétricas introduciendo flúor o cromos diferenciales acoplados a cada uno de los termina dores y sobre todo diseñando equipos automatizada en el proceso con estas nuevas incorporaciones y sobre todo con la automatización se pudo abordar la secuenciación de fragmentos de gran tamaño genomas para la secuenciación de estos fragmentos se suelen utilizar básicamente dos tipos de aproximaciones o algunas aproximaciones híbridas entre ellas por una parte está la aproximación conocida como el software clonación o basada en mapas que conlleva es una fase de pre secuenciación en la que se identifican mediante marcadores moleculares y se ordenan toda una serie de clones solá partes como aparecen en la figura estos clones son los que son secuencia 2 y luego como se sabe el orden en el que ellos están presentes se reconstruye la secuencia ensamblada la otra aproximación se conoce como el software no aplicado al genoma completo secuenciación del genoma completo y en este caso el genoma completo se fragmenta al azar se clona y se secuencia en directamente los fragmentos sin tener ese conocimiento previo de su de su disposición sólo la parte entre ellos nadie se conoce en las secuencias de los fragmentos secuencia 2 estos se reconstruyen en una única secuencia mediante procesos bioinformáticos que analizan dichas secuencias este método estos métodos son los que permitieron establecer elaborar el genoma humano y los genomas de las especies modelos que se desarrollaron a lo largo de principios de los años 2000 pero en el año 2005 aparecen toda una nueva serie de tecnologías parece una revolución tecnológica en la que se presentan los que se conocen como tecnologías de nueva generación que permiten obtener millones de bases de datos en millones de bases en un corto periodo de tiempo estas tecnologías se basan en llevar a cabo la secuenciación en una superficie muy reducida en los datos obtenidos a partir del análisis de imagen de estas reacciones necesitando un tratamiento informático adecuado por ejemplo la empresa roche 454 fue la primera en comercializar uno de estos sistemas y su plataforma utiliza una serie de microesferas que cortan unos fragmentos de adn a secuenciar cada fragmento de estos es amplificado por pcr con lo cual cada micro esfera como tal constituyen una reacción de d de secuenciación la reacción en concreto se lleva a cabo por síntesis con detección de un indigente o pirosecuenciación y consiste en que el nucleótido que hay presente en él en el medio es incorporado por la polimerasa como núcleo complementario a la secuencia molde esta incorporación genera un kilo fosfato que es metabolizado por una sulphur y la ssa y por una luciferasa de manera que aparece un destello luminoso como consecuencia de la incorporación del nucleótido sea bien incorporación de un nucleótido de estudio luminoso la lectura de estos destellos luminosos es la que conlleva el saber que el nucleótido ha sido secuencia ha sido introducido en cada en cada momento otra empresa ilumina ha desarrollado la plataforma solex a que utiliza una estrategia diferente en esta se utilizan determinadores reversibles los terminador es reversibles lo que hacen es que la secuencia a la el fragmento secuencial se fija sobre una superficie sólida mediante unos adaptadores luego es amplificado generando unas regiones unos clases en los que todos los fragmentos que se encuentran en esa zona son idénticos y sobre esa clase sobre esa región se va a producir la reacción de secuenciación en este caso a cada uno de estos fragmentos se le une un observadores y se añaden los primeros nucleótidos cada uno con un determinado flúor o cromo con una determinada marca de fluorescencia los nucleótidos se unen por complementariedad de bases con la secuencia con el flamente a secuencial cuando están unidos se excita el flúor o cromo posteriormente se eliminan los fluor o cromos y los terminador es que están bloqueando la reacción de polimerización y se añaden nuevos nucleótidos con el correspondiente fuego cromo para hacer una segunda lectura de cada uno de los puntos de secuencia con ello se consigue de manera simultánea miles de agrupar agrupamientos de secuencias de adn cada uno correspondiente a una a un fragmento distinto lo que permite obtener gran cantidad de secuencias en un corto periodo de tiempo aún así actualmente ya se ha generado una tercera generación de técnicas de secuenciación que lo que pretenden es secuenciar moléculas únicas en tiempo real sin tener que requerir la amplificación coronal de la molécula como era en los casos anteriores entre éstas vamos a destacar lo vamos a comentar la plataforma amina jon que es basada o desarrollada durante esa jornada por technologies que se basa en la secuenciación de moléculas únicas muy bien que la detección electrónica está a su paso de esta cuadrículas por nano por un nano por o es un agujero de tamaño nanométrico que puede ser creado de manera sintética bien por proteínas en forma de embudo o bien como agujeros en materiales sintéticos y la técnica consiste en asociada a este enano por una enzima que lo que hace es obligar al adn que pase por el nano poro pero qué pasa en base a base de forma que cuando estas bases atraviesan el nano poro el campo eléctrico que hay en el mismo se ve distorsionado de una forma concreta cada nucleótido cada base distorsiona el campo eléctrico de una manera concreta con lo que da una firma eléctrica específica y esto es recogido por el equipo informático con lo cual va leyendo en tiempo real cada una de estas de estas incorporaciones para determinar comentaremos que el presión de secuenciar un genoma completo hoy en día se ha visto muy reducido respecto al precio que significó secuenciar el genoma humano por primera vez y aunque hay muchos parámetros a definir y matices a considerar para lo que puede costar secuenciar un genoma así que podemos decir que con estas tecnologías a finales del 2015 la secuenciación de un genoma como el genoma humano podría podría conseguirse por 1500 dólares incluso por 1000 dólares y lo que queremos secuenciar es el exoma es decir el conjunto de genes de regiones que están funcionales del genoma muchas gracias por vuestra atención [Música]